Dispositivo inspirado na molécula que armazena nosso código genético levará à miniaturização de mídias e garantirá a integridade das informações por muito mais tempo
Por Domingos Zaparolli em Pesquisa Fapesp | A explosão de dados gerados no mundo, por diferentes equipamentos e nos mais diferentes contextos, tem levado cientistas a pesquisar o uso de versões sintéticas do DNA para guardar informações digitais. O DNA, ou ácido desoxirribonucleico, é o sistema de armazenamento de dados da maioria dos seres vivos. É uma molécula presente em todas as células e que carrega as informações genéticas de um organismo. Graças ao estudo de materiais genéticos como o DNA preservado na natureza, temos acesso às informações biológicas dos neandertais, hominídeos extintos há mais de 30 mil anos, e de mamutes, que viveram mais de 1 milhão de anos atrás.
O potencial dos benefícios dessa nova tecnologia é significativo. Segundo a DNA Data Storage Alliance, associação que reúne empresas globais de tecnologia com o objetivo de impulsionar o ecossistema de pesquisa e inovação da técnica, a capacidade de armazenamento de dados em DNA é 115 mil vezes superior à das mídias magnéticas empregadas atualmente nos centros de processamento de dados, os chamados data centers. No mesmo espaço físico de um cartucho de fita magnética LTO-9, capaz de armazenar 18 terabytes (TB), ou seja, 18 trilhões de bytes, é possível guardar em DNA cerca de 2 milhões de TB.
“O data center do Facebook no Oregon, nos Estados Unidos, ocupa uma área estimada de dezenas de milhares de metros quadrados [m²], o equivalente a um grande shopping center, para armazenar uma quantidade de dados da ordem de 1 milhão de TB. O mesmo conteúdo poderia ser armazenado em apenas 5 gramas de DNA, em um dispositivo que cabe na palma de uma mão”, compara o engenheiro eletricista Bruno Marinaro Verona, gerente do Laboratório de Micromanufatura do Instituto de Pesquisas Tecnológicas (IPT) de São Paulo. Verona lidera um projeto de pesquisa na área no Brasil.
Além da densidade, o armazenamento em DNA reúne outros atributos importantes. “É um sistema ambientalmente sustentável”, destaca o engenheiro eletricista paulista Luis Ceze, professor da Escola Paul G. Allen de Ciência da Computação e Engenharia da Universidade de Washington, nos Estados Unidos. Os data centers são intensivos em consumo de energia para a manutenção dos equipamentos e climatização adequada das salas onde são mantidos os arquivos em discos rígidos (HD) e as fitas magnéticas. O DNA, no entanto, pode ser mantido em temperatura ambiente. Além disso, as mídias magnéticas atuais são produzidas com insumos provenientes de mineração de terras-raras e derivados de petróleo e demandam substituição periódica, em no máximo 30 anos. Os pesquisadores estimam que os dados digitais arquivados em DNA serão legíveis por milhares de anos.
A evolução dos processos de arquivamento de dados digitais é necessária diante da gigantesca quantidade de informações digitalizadas geradas com a expansão do uso da tecnologia da informação (TI), ou seja, o uso de computadores e smartphones para criar, processar e trocar todo tipo de dado. Segundo relatório da consultoria norte-americana IDC, em 2010 foram gerados no mundo 3 zettabytes (3 seguido por 21 zeros) de dados novos e backups, as chamadas cópias de segurança. Em 2020, esse total saltou para 64 zettabytes (ZB) e a projeção é alcançar 180 ZB em dois anos.
Como registra a DNA Data Storage Alliance, esse é apenas o começo do que vem sendo chamado de era da informação, na qual a inteligência artificial e a internet das coisas estarão cada vez mais presentes em todas as atividades do dia a dia das pessoas, da saúde à educação, do comércio à condução de um veículo ou à operação de uma fábrica. De acordo com a associação, apenas um carro autônomo gera 15 TB de dados brutos por período de oito horas. Nem todos esses dados são arquivados, mas parcela significativa é mantida para demandas diversas, como segurança pública e manutenção automotiva.
Hoje esses dados são arquivados utilizando arquiteturas remotas de armazenamento baseadas em nuvem. “Os data centers consomem cerca de 1% da energia elétrica produzida no mundo. A indústria de TI prevê que o consumo passará para 30% da energia global em poucos anos”, alerta Hildebrando Lima, diretor de Pesquisa e Desenvolvimento da Lenovo no Brasil. “É preciso criar uma alternativa que reduza esse impacto, e a técnica de armazenamento em DNA é a aposta mais promissora.”
Cientistas envolvidos no desenvolvimento da metodologia de armazenamento em DNA reconhecem que a solução vai demorar para estar disponível ao público. As projeções apontam para a próxima década. Ainda há incertezas em relação aos processos que serão utilizados. “O que podemos afirmar é que o armazenamento de dados digitais em DNA é viável”, afirma a engenheira eletricista brasileira Karin Strauss, gerente sênior de pesquisa da Microsoft Research, em Redmond, nos Estados Unidos. “Já produzimos em laboratório e, ao que tudo indica, parece ser possível realizar o processo em escala comercial de forma econômica, mas ainda temos muito o que avançar para chegar a esse objetivo.” Uma das fundadoras da DNA Data Storage Alliance, a Microsoft Research é o laboratório de pesquisa da multinacional de TI.

A produção de DNA sintético e o armazenamento de dados nele já são realizados pela indústria de biotecnologia para uso na área da saúde, mas a escala produtiva é baixa e a velocidade do processo é lenta diante das necessidades da indústria de TI. “Hoje é possível escrever dezenas de megabytes [MB] por segundo em um HD magnético convencional. Para guardar um único MB em DNA levamos um dia de trabalho”, compara Verona, do IPT. “Todas as pesquisas têm o mesmo objetivo: estabelecer as melhores técnicas de armazenamento de dados em DNA e aprimorá-las”, complementa Ceze, da Universidade de Washington.
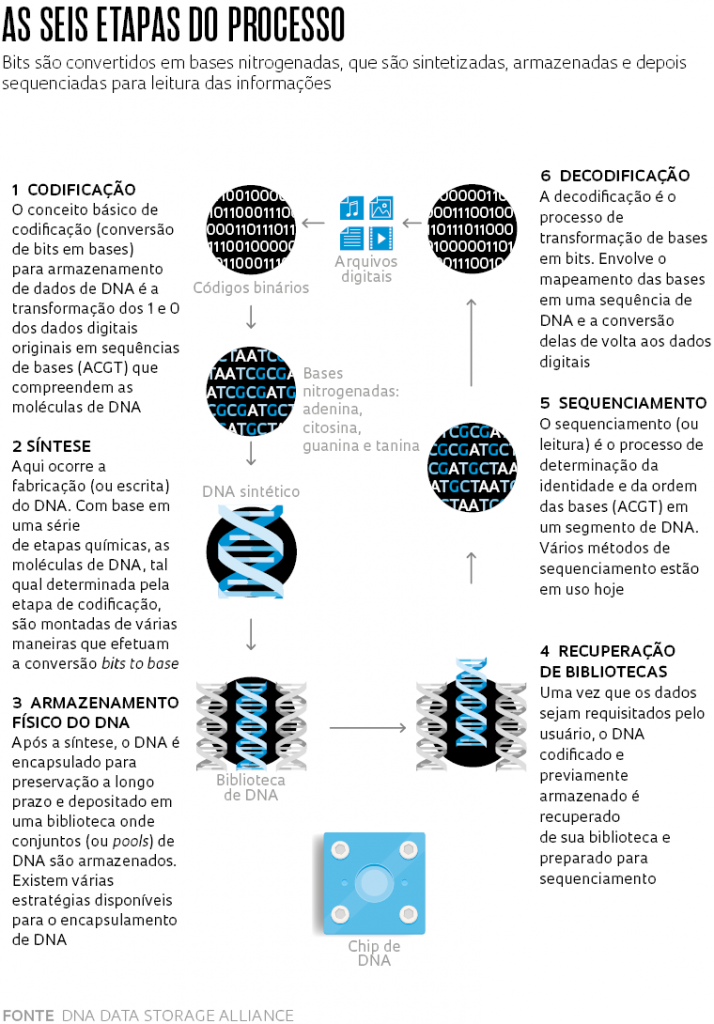
O processo para armazenar dados em DNA não envolve manipulação genética ou de células de organismos vivos. O DNA é fabricado por meio de síntese química e cada molécula é construída na medida exata em que os arquivos de dados são gerados. Isso significa que para guardar um 1 TB de informações será preciso sintetizar um conjunto de moléculas com essa capacidade de armazenamento (ver infográfico).
Esses novos dispositivos serão constituídos de estruturas capazes de sintetizar e sequenciar as fitas de DNA, que contêm as informações digitais de interesse. Um dos designs possíveis é que ele contenha microcavidades, de alguns nanômetros de profundidade, em que as moléculas de DNA seriam sintetizadas. A primeira etapa da fabricação do arquivo é nomeada pelos cientistas de bits to base. Consiste em converter por meio de programas computacionais os bits – o sistema binário 1 e 0 usado na computação para representar caracteres, números e imagens – nas quatro bases nitrogenadas que compõem a molécula de DNA: adenina (A), timina (T), citosina (C) e guanina (G) (ver Pesquisa FAPESP no 235). Na molécula, as bases nitrogenadas têm a função de formar os dois filamentos em espiral, conhecidos como dupla hélice, que constituem o DNA.
Para exemplificar: os bits 00 podem ser codificados como base A; os bits 01 como base T; os bits 10 como C; e os bits 11 como G. Quando se quer acessar os dados e ler os arquivos, é só fazer a decodificação, ou seja, converter a base nitrogenada em bits. No jargão do setor, fazer o base to bits. As informações armazenadas em DNA poderão ser acessadas via internet ou localmente. A conversão de bases para bits é feita por um processo computadorizado e rápido, embora ainda mais lento do que o tempo de leitura de arquivos magnéticos tradicionais.
O principal desafio a ser solucionado pelos pesquisadores é melhorar os métodos de síntese química empregados para escrever os códigos em bases nitrogenadas e construir simultaneamente as moléculas sintéticas de DNA.
Existem dois processos estabelecidos pela bioengenharia. O mais antigo é a síntese química de fosforamidita, criada nos anos 1980 pelo bioquímico norte-americano Marvin H. Caruthers. É o processo predominante para aplicações em atividades biomédicas. O outro caminho possível é a síntese enzimática, que vem sendo aperfeiçoada por vários grupos de pesquisa nos últimos 15 anos, mas ainda não chegou ao estágio comercial.
A síntese enzimática utiliza moléculas orgânicas proteicas que trabalham como catalisadoras das reações químicas, ou seja, aceleram a velocidade dos processos. Uma vantagem desse processo é o uso de reagentes aquosos, não tóxicos. Dessa forma, gera menor impacto ambiental quando comparada com a síntese por fosforamidita, que utiliza reagentes fósseis. Para Ceze, a síntese enzimática, tecnologia ainda no nascedouro, tem maior potencial de evolução e deverá prevalecer.
A Microsoft pesquisa tecnologias de armazenamento em DNA desde 2015 em parceria com o Laboratório de Sistemas de Informação Molecular da Universidade de Washington. Entre os vários estudos conjuntos que geraram artigos científicos, Strauss destaca dois. Em 2019, um paper publicado na Nature Scientific Reports indicou a viabilidade de automação da síntese química, eliminando o trabalhoso processo de pipetagem (a transferência de líquidos) manual de DNA, como é realizado ainda hoje. “A automação irá gerar escala e reduzir os custos do processo de armazenamento em DNA”, projeta a pesquisadora.

Em 2021, outro artigo conjunto das duas instituições publicado na Science Advances apresentou um sistema de gravação de DNA em nanoescala que usa um método de controle do ambiente molecular capaz de permitir a geração ao mesmo tempo (em paralelo) de um grande número de sequências de DNA exclusivas. “O resultado apresentado nesse trabalho foi a miniaturização da unidade de escrita de uma sequência e do processo químico que a controla, de forma que caibam mais dessas unidades no mesmo chip”, destaca Strauss. As fitas hoje não superam 300 bases nitrogenadas, o que representa registrar menos de 30 bytes (o conjunto de oito bits) por sequência.
No Brasil, o único grupo de pesquisa que integra a DNA Data Storage Alliance resulta de uma parceria entre o IPT e a fabricante chinesa de dispositivos eletrônicos Lenovo. Batizado de Prometheus, o projeto teve início em 2021 e é coordenado por Verona. Fazem parte da equipe multidisciplinar 40 pesquisadores, sendo 13 mestres e 21 doutores entre biólogos, engenheiros da computação, moleculares, químicos e de materiais. O IPT é o único parceiro global da Lenovo no desenvolvimento de tecnologias de armazenamento em DNA.
De acordo com Lima, da Lenovo, a parceria produziu quatro depósitos de patentes internacionais, sendo uma resultante do trabalho da equipe de codificação e decodificação dos dados, duas em sintetização química e uma em sintetização enzimática. “Temos outras seis pesquisas no forno e em breve chegarão ao estágio de pedido de patente”, revela o executivo.
Verona pondera que a tecnologia de arquivamento de dados em DNA deverá ter um avanço gradual e, em um primeiro momento, não deverá estar disponível em computadores e smartphones. Inicialmente, as soluções serão direcionadas ao chamado armazenamento frio, composto de dados que os usuários não acionam rotineiramente em seu dia a dia — uma espécie de arquivo morto digital —, como históricos de todos os tipos, álbuns de fotos e vídeos. Nos data centers atuais, esses arquivos são armazenados em fitas magnéticas.
Os dados acessados frequentemente, os quentes, hoje são arquivados em HD. “Ainda não há uma projeção confiável de quando o arquivamento em DNA será capaz de se apresentar como alternativa ao armazenamento de dados quentes”, diz Verona. Para Ceze, vários sistemas de armazenamento de dados deverão conviver simultaneamente no futuro, possibilitando o melhor aproveitamento de suas respectivas características para cada finalidade.
Este texto foi originalmente publicado pela Pesquisa Fapesp de acordo com a licença Creative Commons CC-BY-NC-ND. Leia o original. Este artigo não necessariamente representa a opinião do Portal eCycle.